Installation (French)
Cette partie du Wiki explique comment installer le logiciel fournisseur de données BioCASe (‘BioCASe Provider Software’ aussi appelé "BPS"). Cela devrait prendre entre 15 et 60 minutes. Si vous êtes un débutant, veuillez lire le Guide du Débutant Beginner’s Guide en premier lieu.
Prérequis
Avant de commencer l’installation, assurez-vous que le système sur lequel vous avez l'intention d'installer le BPS remplit les conditions suivantes:
- Vous avez des privilèges d'administrateur sur cette machine, c'est à dire les droits d'utilisateur pour installer de nouveaux logiciels.
- Il est accessible de l'extérieur de votre établissement. Cela signifie qu'il doit soit être à l'extérieur d'un pare-feu institutionnel ou que, plus probablement, le port HTTP pour un accès Web est ouvert pour un pare-feu existant [note : il n’est pas nécessaire de staisfaire cette condition si vous installez BioCASe a des fins de test ou sur votre propre ordinateur pour vous entraîner individuellement].
- Une connexion directe peut être établie à partir de cette machine à la base de données à publier - soit parce qu'il n'y a pas de pare-feu entre les deux, ou bien que le port utilisé par le SGBD est ouvert (par exemple 3306 pour MySQL, 5432 pour PostGres, 1433 pour Microsoft SQL server). Si le logiciel BioCASe est installé sur la même machine que le serveur de base de données, le pare-feu n’a normalement pas à être ouvert.
Si une de ces conditions n'est pas remplie, veuillez contacter votre administrateur système pour de l'aide, lisez la FAQ ou prenez contact avec l'équipe BioCASe.
Installation de Apache / IIS et Python
Si vous utilisez un système Linux, vous n'avez probablement pas besoin d'installer ces logiciels, car Apache et Python sont généralement préinstallés. Il en va de même pour MacOS X, mais vous devriez malgré tout jeter un oeil aux discussions et à la documentations accessible sur le web pour obtenir plus d'information sur les emplacements de fichiers dans le système.
Même si le provider fonctionne avec Apache et Internet Information Server (IIS) de Microsoft , nous vous recommandons d'utiliser Apache, qui est beaucoup plus facile à configurer. Toutefois, si vous utilisez déjà IIS, vous souhaitez peut-être le conserver. Les versions d'Apache recommandées sont 2.0 et 2.2.
Le BPS nécessite la version 2.5 de Python afin de fonctionner. Même s’il peut fontionner également avec les versions 2.6 et 2.7, la version recommandée est de 2.5 Python (en fait la dernière sous-version stable de la branche 2.5: 2.5+). Pour les versions ultérieures, les librairies nécessaires pour la connexion du BPS à la base de données utilisé ne sont peut être pas encore disponible. Vous pouvez aller à la page de test des librairies de l'installation du Musée et Jardin Botanique de Berlin pour vous procurer le paquet d'installation du driver de votre base de données, et vérifier sa disponibilité pour de nouvelles versions de Python sur le bib site Web (par exemple, la bibliothèque 4SuiteXML utilisée pour l'exécution de l’interface de requête locale est actuellement uniquement disponibles pour Python 2.5). Soit dit en passant: vous pouvez installer plusieurs versions différentes de Python sur la même machine sans conflits.
Si vous faites l’installation sur un ordinateur Windows Vista ou Windows 7, nous vous recommandons de ne pas installer Apache dans le dossier Program Files par défaut. En effet à cet endroit le serveur Apache a un comportement imprévisible qui peut vous causer des ennuis lors de la modification de fichiers de configuration. Il suffit de choisir un autre chemin (par exemple C:\\Apache22 ou D:\\Apache22) pour parer à cet inconvénient.
Dès que vous avez installé avec succès Apache et Python, vous pouvez procéder à l'étape suivante.
Téléchargement du logiciel fournisseur
Vous pouvez télécharger le BPS soit comme une archive zippée g-ou directement à partir du Subversion du BGBM (Subversion ou SVN est un logiciel qui permet à plusieurs programmerus de travailelr en parallèles sur le même code, il facilite aussi l'archivage des versions du code et la docuemntations des changements en les stockant sur un réseau). La première option est la plus facile si vous n'êtes pas familier avec Subversion. Avec la deuxième option, il sera plus facile de mettre à jour votre installation ultérieurement. Si vous n'avez jamais utilisé Subversion avant et ne prévoyez pas de mise à jour régulière de votre installation, choisissez l'archive. Mais si vous prévoyez de mettre en place une installation de production, qui devrait être mise à jour, vous devriez opter plutôt pour le dépôt Subversion.
Téléchargement du fichier d'archive
Il suffit de télécharger l'archive à partir de la partie Téléchargement du site BioCASe et de décompresser le contenu du dossier de destination.
Téléchargement à partir du dépôt Subversion
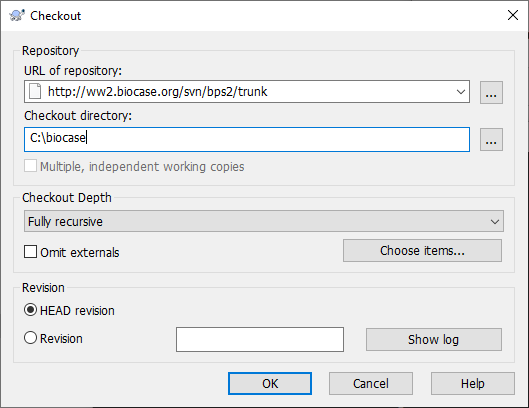
Si vous utilisez Windows, vous devez installer un client Subversion premier (vous pouvez essayer Tortoise, qui est livré avec une jolie interface très commode). Linux et MacOS contiennent habituellement un client Subversion préinstallé.
L'URL de la dernière version stable est http://ww2.biocase.org/svn/bps2/branches/stable, que nous vous recommandons d'utiliser. La mise à jour de votre installation avec la dernière version BPS est alors facile, et revient à exécuter la commande « update » de Subversion. Si vous voulez télécharger une version définie, l'URL serait (exemple dans le cas de la version 2.6.0) http://ww2.biocase.org/svn/bps2/tags/release_2.6.0 .
Pour télécharger le BPS avec le client Subversion en ligne de commande (préinstallé sur Linux / MacOS), allez simplement au répertoire sur lequel vous voulez décompressez l’archive et introduisez :
svn co http://ww2.biocase.org/svn/bps2/branches/stable biocase
Vous vous connectez alors au serveur du Jardin et Musée Botanique de Berlin avec un accès anonyme et télécharger tous les fichiers du sous-répertoire biocase. Dans le cas où vous utilisez Tortoise sous Windows, faites ub clic-droit sur le répertoire de destination dans l'explorateur Windows et choisissez «Checkout». Introduisez l'URL (http://ww2.biocase.org/svn/bps2/branches/stable), changez le nom du dossier de destination sur votre machine pour qu’il soit significatif et facilement reconnaissable par vous et appuyez sur OK:
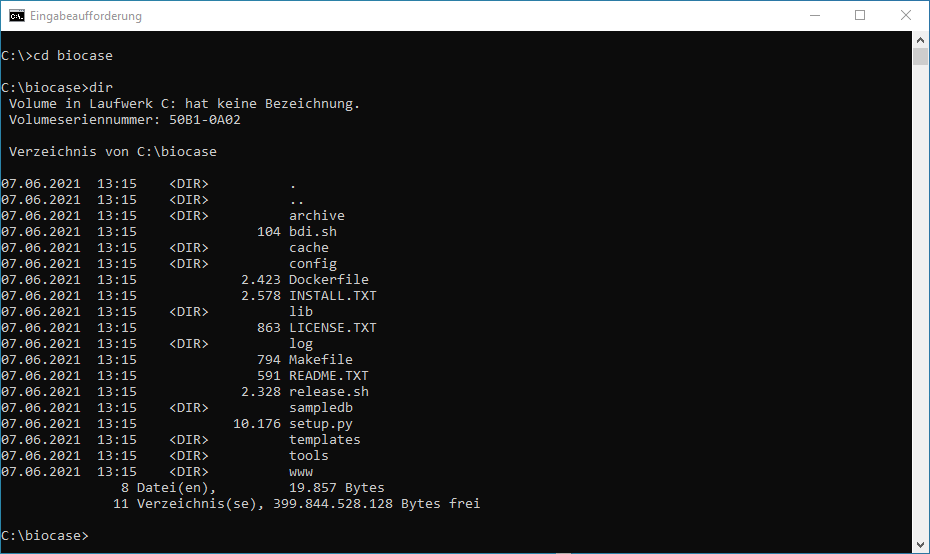
Nous supposerons dans la suite de cette explication que vous avez installé le logiciel BPS dans un dossier nommé BioCASe, qui devrait ressembler à ceci maintenant:
Exécution du script d'installation
Le script d'installation va paramétrer les fichiers de configuration avec les valeurs que vous lui fournissez (en fonction de votre installayion) et lier les fichiers de script à votre installation de Python. Vous pouvez ré-exécuter le script de configuration dans le cas où vous modifiez l’emplacement d’Apache, Python ou du logiciel BioCASe lui-même sur votre ordinateur. Si vous souhaitez modifier ou corriger l'un des paramètres que vous avez déclarés pendant l'installation, vous pouvez également changer les valeurs en relançant à nouveau le script d’installation.
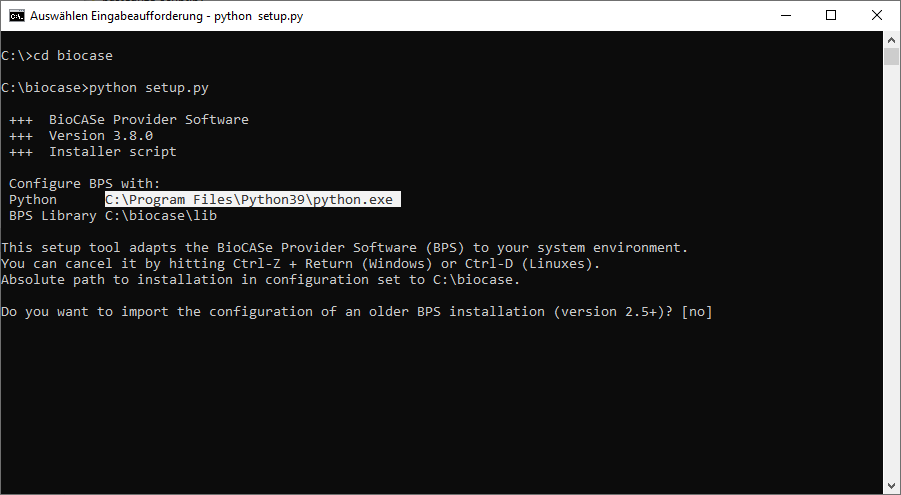
Pour exécuter le script, ouvrez une fenêtre de terminal (appelée «invite de commande", "commandes MS-DOS" ou encore "command prompt" en anglais dans Windows) à laquelle vous pouvez accéder sur Windows via le sous-menu « accessoire » du menu général, ou bien en tapant «cmd» dans le menu « exécuter ». Changez le répertoire de travail vers le dossier que vous avez téléchargé le BPS (par exemple si votre provider est installé à l'emplacement D:\\biocase
D:
cd D://biocase
. Tapez ensuite:
python setup.py
Si la commande ne marche pas, vous devez peut-être ajouter le nom de chemin complet vers l'exécutable Python pour que la fenêtre d'installation puisse le trouver. Exemple sous Windows et DOS, en supposant que vous avez installé Python dans sa version 2.5 et dans le répertoire C:\Python25:
C:\Python25\python.exe setup.py
...adaptez les valeurs en fonction de la situation sur votre ordinateur) Assurez-vous que la version de Python donnée dans la cinquième ligne de la sortie correspond à celle que vous désirez (sinon ajoutez le chemin complet vers la bonne version de l’intepréteur comme montré ci-dessous):
Les questions posées par le script de configuration proposent des valeurs par défaut. Si vous souhaitez les conserver, il suffit d'appuyer sur Entrée.
Webserver domain: Il s'agit du nom de domaine du serveur Web que vous installez sur BioCASE, par exemple, http://www.moninstitution.org. Vous pouvez conserver la valeur par défaut localhost tant que vous configurez et testez l'installation localement, mais pour une machine la production réellement accessible par Internet, vous aurez besoin de changer cela dans le nom de domaine correct (ou adresse IP) ultérieurement. This is the domain name of the web server you’re installing BioCASe on, e.g. http://www.myinstitution.org.
Base URL: Ce sera la deuxième partie de l'URL de votre installation (l’adresse du site dans le serveur, par exemple http://moninstitution.org/monbiocase/ ). Si vous gardez la valeur par défaut, votre installation sera accessible plus tard à www.myinstitution.org/biocase. Ce paramètre correspond à une option des paramètres Apache.
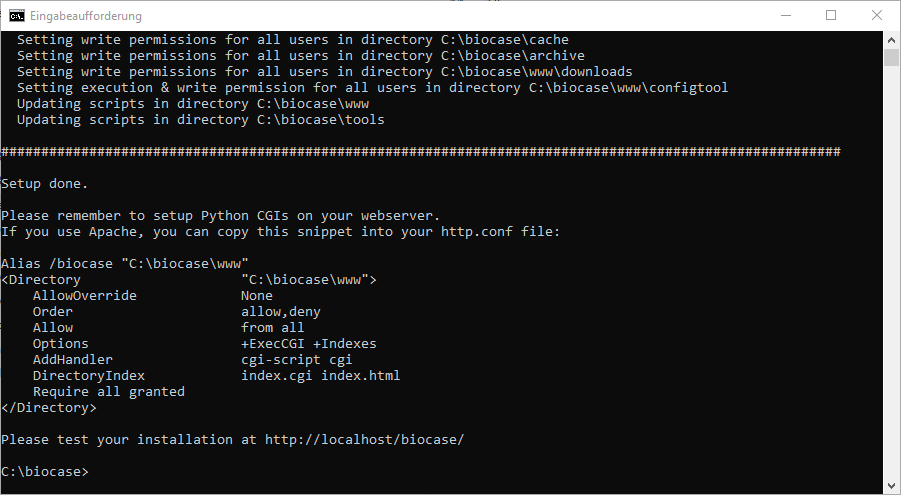
Après avoir répondu à ces deux questions, le script mettra à jour vos fichiers. À la fin de la sortie que vous pouvez trouver les lignes dont vous aurez besoin plus tard pour créer le lien entre BioCASe et votre serveur web. Il ne faut donc pas fermer la fenêtre!
Configurer votre serveur Web pour BioCASe
Les fichiers et programmes visibles publiquement depuis le Web sont placés dans le sous-répertoire www de votre installation BioCASe et doivent être configurés sur votre serveur pour un accès en lecutre depuis l'extérieur. Vous devriez toujours configurer cela à l'aide d'un répertoire virtuel: c'est à dire que vous ne devez pas installer BioCASe à la racine de votre serveur Web (par exemple sur '/var/www' sur Ubuntu). Sinon vous pouvez compromettre la confidentialité de votre serveur et permettre au modne extérieur de voir vos fichiers de configuration (qui incluent des mots de passe). Ainsi si vous avez installé BioCASe sur d:\biocase, le répertoire que vous devez déclarer comme répertoire virtuel dans la configuration de votre server est d:\biocase\www' et sera accessible par Internet via http://www.moninstitution.org/biocase.
Apache
Si vous utilisez Apache, l'ajout du répertoire BioCASe dans votre environnement a lieu simplement en copiant les directives affichées à la fin du script d'installatioon dans le fichier de configuration d'Apache (httpd.conf dans le cas de la plupart des installations sur Windows). Ce fichier est le plus souvent situé dans le sous-répertoire conf de votre installation d'Apache. Si vous installer BioCASe sur un serveur qui est déjà utlisé pour publier des sites webs, veuillez en informer l'administrateur avant de modifier ce fichier!
Sauvee le fichier et redémarrez Apache pour mettre en vigueur les modifications (sous Windows ce redémarrage est souvent possible via l'interface graphique de votre Apache ou du package WAMP que vous avez installés, sous Linux via une commande Shell, comme sous Ubuntu
sudo /etc/init.d/apache2 force-reload
.
Internet Information Server (IIS)
Si vous utlisez IIS vous devez créer d'abord un répertoire virtuel pour le répertoire physique c:\biocase\www en utilisant l'outil IIS Server Manager. Allouez tous les droits pour ce répertoire (Read, Write, Script Execution, Directory listing), utilisez le bouton Configure sur l'onglet virtual directory et associez finalement les extensions de fichier .cgi and .py avec le chemin de votre fichier exécutable Python. Vous devriez aussi aussi ajouter index.html à la des documents par défaut (Virtual Directory > Properties > Documents > Default).
Nous savons que configurer d'une manière générale Python pour IIS est souvent fastidieux. On trouve néanmoisn de bonenx explciations sur le web, par exemple
- http://www.ecp.cc/pyiis.html
- http://support.microsoft.com/kb/276494
- http://python.markrowsoft.com/iiswse.asp
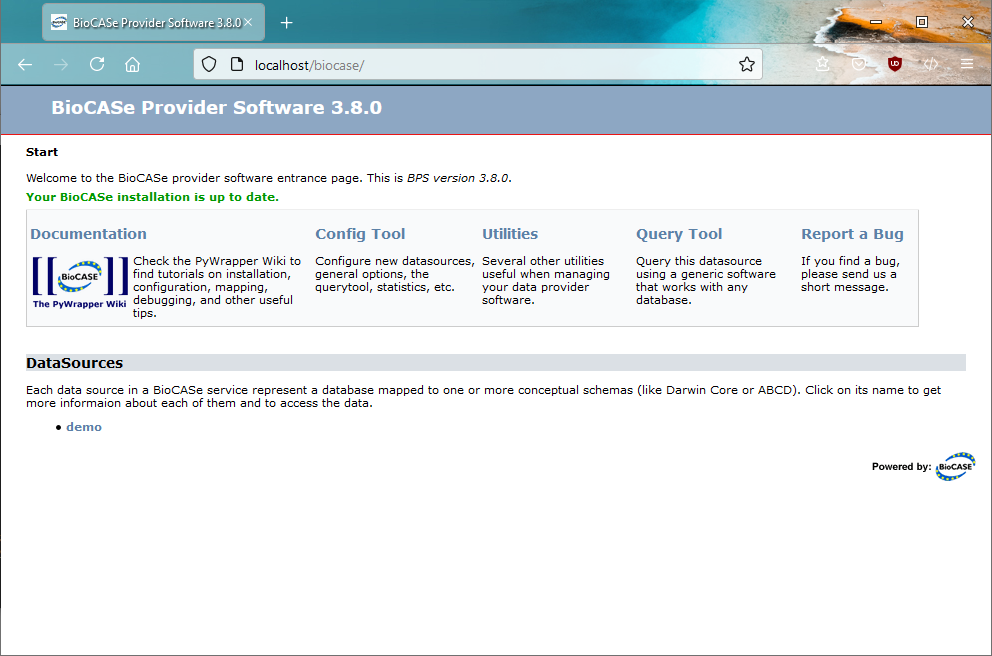
Once you’re done, you can test your BioCASe Installation by opening a browser and typing in http://localhost/biocase (replace localhost with the domain name of the machine you installed BioCASe on, if you’re not working locally). You should then see the start page of the BPS:
Installing Additional Python Packages
Before you can start configuring your installation, you need to install additional packages, depending on the management system used for the database to be published and the functions of the BPS you intend to use.
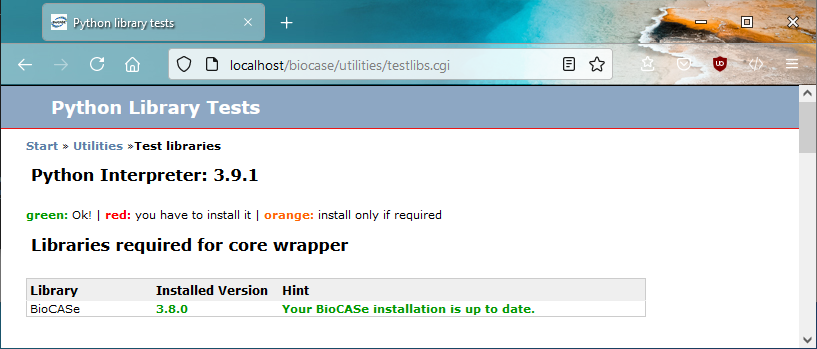
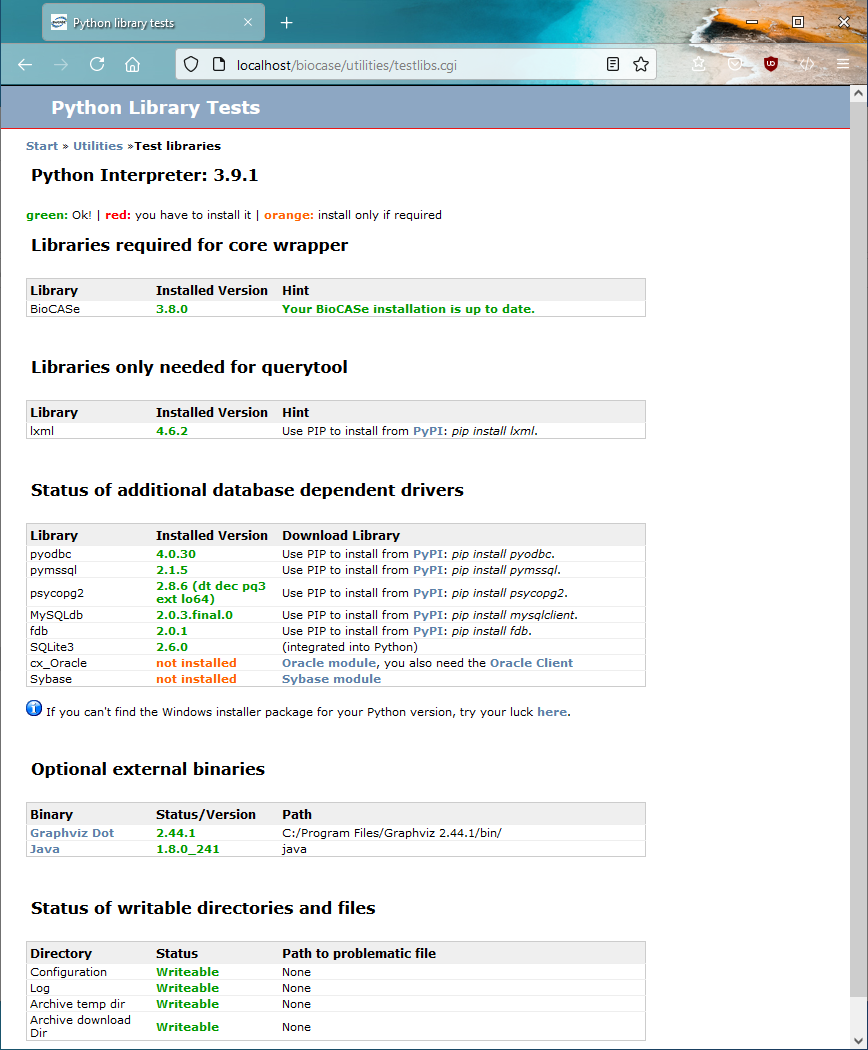
On the BPS start page, click on Utilities, then library test in order to navigate to the library test page that shows the status of installed libraries. On a fresh BPS installation, it should look similar to this:
The BioCASe libraries are already included in the archive file you’ve downloaded (or the Subversion repository you checked out from). The 4SuiteXML library is required only for the Local Query Tool – if you don’t plan to use it, you can ignore it. Otherwise click on the Download link, find the installer appropriate for your operating system and install it.
For each DBMS you plan to use with BioCASe (i.e. you have a database you want to publish with BioCASe), you’ll have to install the corresponding package. So if your collection is stored in a MySQL database, for example, click on the download link for the MySQL module, find the correct installer for your Python version and operating system and install. Once the installation is done, after reloading the libraries test page in the browser, the according list entry should turn from an orange ‘not installed’ into a green version number. Redo this process for all DBMS you want to connect to.
The Graphviz library listed under optional external binaries is used by the BPS to create a graph of your database’s data model in the table setup (you will learn in the Table Setup about this). If the data model is very simple (i.e., not more than 3 or 4 tables), you probably can spare the effort of installing Graphviz. Otherwise we recommend installing it: Simply click on the Download link, find the correct installer, and… you know the story.
Changing the Default Password
The configuration tool is a browser interface that allows you to configure almost every aspect of the BPS. As a last step of the setup, you should change the password.
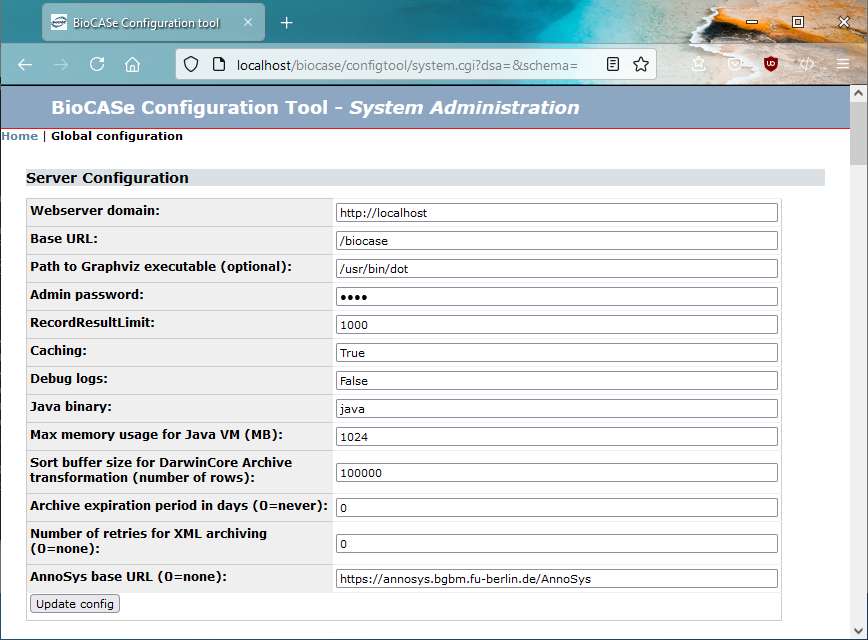
Go back to the Start page of the BPS (by clicking Start at the top of the page, or by retyping http://localhost/biocase if the server is running on your computer), choose Config Tool, then System Administration. You will then be prompted for a password. Type in the default ACDC, then click Submit.
The top of the configuration page you’ll be directed to should look similar to this:
In the first two lines, you’ll see the values you entered during the setup script (in this case, the defaults localhost and biocase). In the fourth line, enter a new password. Please be careful when typing (if you forget the password some day, you can reset it by directly editing the configuration file). Press update config to save the new password to the configuration file.
In case you’ve installed Graphviz, you need to tell the BPS the location of the binaries, since it is not a Python package that will be registered automatically. In order to do so, locate the Graphviz installation on your local disk and enter the path to the binary executable into the text box labelled Graphviz dot binary. On Linux machines it might look similar to the default (/usr/local/bin/dot), on Windows machines it will be probably something like C:\Program Files\Graphviz2.26.3\bin\dot.exe. Press update config to save the path.
Checking Your Installation
Before you proceed to the configuration, you should make sure everything is installed correctly:
- Typing in http://localhost/biocase (or the appropriate values of your installation) takes you to the entry page of the provider software (as shown in the section Linking BioCASe and Your Web Server).
- The libraries test page (Utilities > library test) shows you’ve installed the packages for you DBMS, 4SuiteXML (if you want to use the Local Query Tool) and the Graphviz Dot (if you decided so).
- Status of writeable directories shows a green writeable for both the Logs and the Configuration. If not, the path to problematic file will be given – please grant write access to Python.
Let’s assume you want to use BioCASe in conjunction with a Postgres and an Access database (which will be connected to through ODBC) and you don’t care for the Query Tool, the libs page should look like this: